Naukowcy z Laboratorium Biomolekularnych Interakcji i Transportu UAM/MIBMiK, kierowanego przez dr Jana Brezovskiego, niedawno opublikowali artykuł zatytułowany „TransportTools: a library for high-throughput analyses of internal voids in biomolecules and ligand transport through them”.
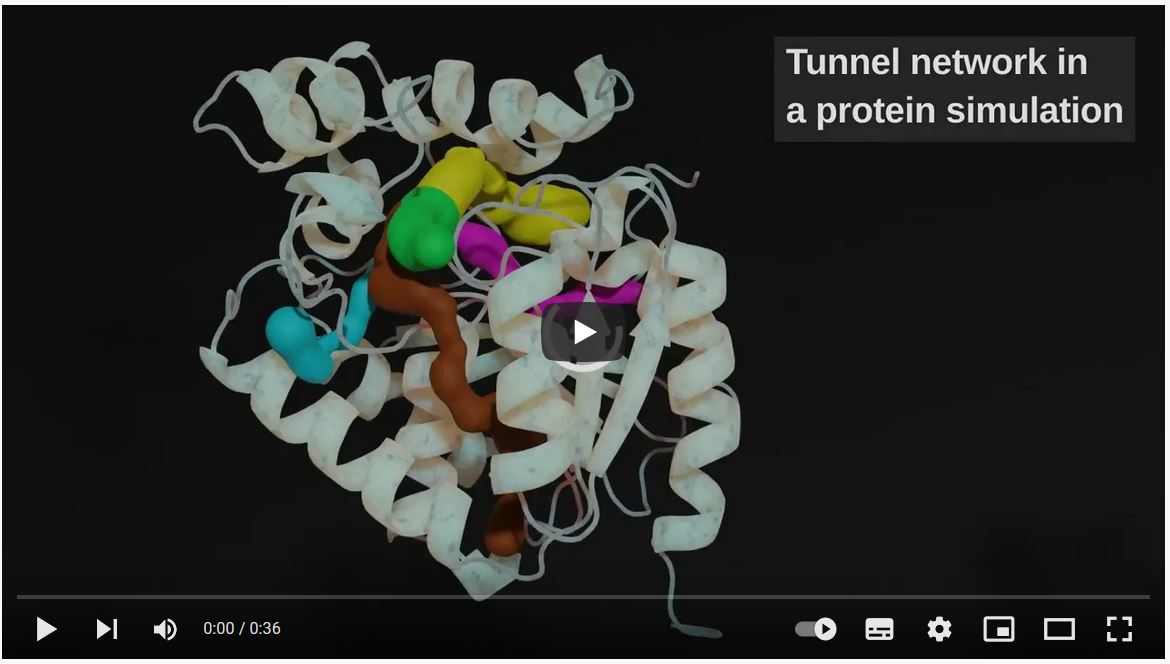
Transport małych cząsteczek organicznych w obrębie komórki i poza nią ma kluczowe znaczenie dla utrzymania homeostazy. Transport ten jest zapewniony głównie przez kanały i tunele utworzone z wewnętrznych pustych przestrzeni biomolekuł. W związku z tym tunele i kanały są kluczowym celem w projektach odkrywania leków i inżynierii białek. Niestety, szlaki te są często wyposażone w dynamiczne bramy, co czyni je przejściowymi i trudnymi do zbadania. Jednym z najbardziej efektywnych sposobów scharakteryzowania tych sporadycznych przypadków transportu ligandów przez przejściowo otwarte szlaki jest przeprowadzenie symulacji dynamiki molekularnej (MD). Nierzadko trzeba wykonać tysiące symulacji, aby uchwycić to sporadyczne zdarzenie, kiedy ścieżka otwiera się i pozwala ligandowi przejść. Jednakże, takie wysokoprzepustowe metody stanowią znaczne wyzwanie dla badaczy związane z ustaleniem tożsamości ścieżek obserwowanych we wszystkich symulacjach, określeniem, które ścieżki są wykorzystywane przez poszczególne ligandy oraz opracowaniem środków do specyficznych analiz ilościowych.
W tym celu przedstawiamy TransportTools: bibliotekę zaprojektowaną w celu zredukowania tych trudności poprzez zapewnienie łatwego i efektywnego dostępu do szczegółowych informacji na temat procesów transportu, nawet w przypadku dużych zestawów symulacji, oraz zaoferowanie środowiska do opracowywania nowych analiz i narzędzi.
Biblioteka ta jest przydatna w następujących przypadkach:
-
analiza ścieżki transportu w rozległych symulacjach MD, w tym pochodzących z masowo równoległych obliczeń lub bardzo długich symulacji;
-
ponowna integracja danych dotyczących szlaków transportu i ich rzeczywistego wykorzystania przez małe cząsteczki, w miarę możliwości z ogromnego zbioru symulacji;
-
porównanie procesów transportu w różnych warunkach, np. przez zestawienie transportu w oryginalnym układzie z tym samym układem zmienionym przez mutacje, różne rozpuszczalniki lub związane ligandy.
Aby zapoznać się z możliwymi zastosowaniami, można prześledzić niektóre z naszych trzech przypadków użycia - dane są dostępne na stronie Zenodo:
przypadek użycia 1) odkrywanie rzadkich tuneli przejściowych i ich wykorzystanie przez cząsteczki wody na podstawie 10 symulacji MD
przypadek użycia 2) zrozumienie wpływu mutacji na system na szlaki transportu poprzez porównanie symulacji typu dzikiego z dwoma mutantami
przypadek użycia 3) określenie selektywności ścieżek dla cząsteczek substratów na podstawie blisko 600 symulacji MD
Narzędzie TransportTools jest zaimplementowane jako moduł Pythona3 i jest dostępne jako pakiety pip i Conda. Kod źródłowy i podręcznik użytkownika są dostępne na GitHubie.
Praca została opublikowana w czasopiśmie naukowym Bioinformatics w 2021 r.