Naukowcy z Laboratorium Struktury Białek IIMCB zbadali struktury polimeraz DNA AbiK i Abi-P2, które biorą udział w obronie bakterii przed inwazją wirusów. Struktury ujawniły bardzo nietypową heksameryczną i trimeryczną architekturę tych enzymów. Na podstawie zestawu struktur i towarzyszących im analiz biochemicznych i biofizycznych wyjaśniono molekularny mechanizm działania polimeraz Abi.
Bakterie wykorzystują szeroki wachlarz strategii obronnych, aby zapobiegać infekcjom wirusowym lub replikacji wirusów. Jedną z nich jest infekcja abortywna (Abi), która jest altruistycznym procesem zaprogramowanej śmierci zainfekowanej komórki w celu uniemożliwienia wirusowi replikacji i rozprzestrzeniania się na inne komórki. Zidentyfikowano wiele systemów Abi i przewiduje się, że mają one bardzo odmienne mechanizmy działania. Białka AbiK i Abi-P2 są białkami efektorowymi Abi, które wykazują podobieństwo do odwrotnych transkryptaz - enzymów, które syntetyzują DNA używając RNA jako matrycy. Wcześniejsze badania biochemiczne AbiK wykazały, że enzym ten może syntetyzować DNA bez użycia matrycy. Co więcej, nie potrzebuje on krótkiego fragmentu DNA do rozpoczęcia syntezy kwasu nukleinowego. Zamiast tego pierwszy nukleotyd rosnącego łańcucha DNA przyłącza do jednej z własnych reszt wykorzystując mechanizm zwany primingiem białkowym.
Naukowcy z Laboratorium Struktury Białek określili pierwsze struktury polimeraz DNA AbiK i Abi-P2 przy użyciu krystalografii rentgenowskiej i mikroskopii krioelektronowej. Oba białka złożone są z dwóch części: katalitycznej domeny podobnej do RT oraz domeny unikalnej dla białek Abi, która zbudowana jest z α-helis i służy do stabilizacji powstającego łańcucha DNA. Struktury ujawniły, że AbiK i Abi-P2 tworzą odpowiednio heksamery i trimery, co jest niespotykane w przypadku polimeraz DNA. Struktura białka AbiK typu dzikiego zawierała jednoniciowy łańcuch DNA, którego koniec 5′ był kowalencyjnie przyłączony do reszty zaangażowanej w priming, zaś koniec 3′ znajdował się w centralnym kanale enzymu. Ponadto za pomocą kriomikroskopii elektronowej określona została struktura wariantu białka AbiK, który zawierał substytucję reszty zaangażowanej w priming, w związku z czym nie posiadał kowalencyjnie związanego DNA. Porównanie struktur z DNA i bez DNA pokazało, że przeprowadzenie primingu białkowego przez AbiK wiąże się ze zmianą ułożenia ruchomej części białka zawierającej resztę aminokwasową biorącą udział w primingu.
Praca została opublikowana w czasopiśmie Nucleic Acids Research
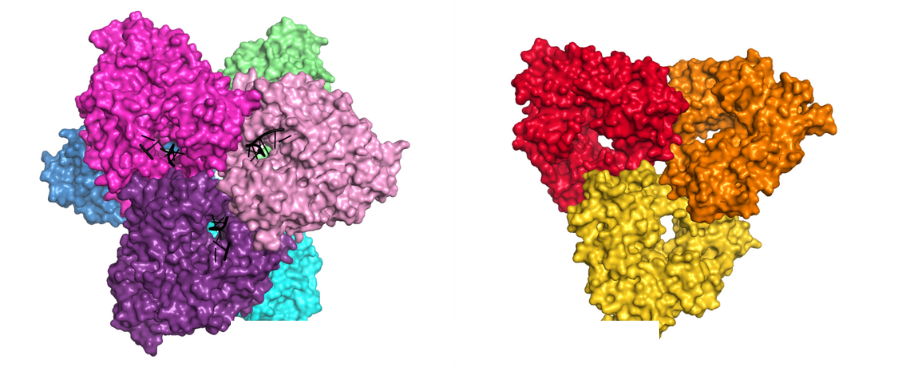
Struktury adduktu złożonego z polimerazy AbiK i DNA (z lewej) oraz polimerazy Abi-P2 (z prawej).
