Pozytywna pamięć genetyczna, pamięć transkrypcyjna – od czego zależy? Jakie dokładnie czynniki za nią odpowiadają? Ostatnio opublikowany artykuł w czasopiśmie Cellular and Molecular Life Sciences autorstwa naukowców z Laboratorium Biologii Strukturalnej przyczynia się do lepszego zrozumienia pozytywnej pamięci genetycznej.
Jak to możliwe, że po podziale komórki (tzw. mitozie) komórka potomna "pamięta" jaką powinna mieć "tożsamość"? Dlaczego komórka nerwowa nie staje się nagle komórką serca, skoro obie mają takie samo DNA? Za prawidłowy mechanizm utrzymania tej "tożsamości" odpowiada pamięć epigenetyczna, która wprowadza odpowiednie znaczniki genów powodujące, że stają się one aktywne lub wyłączone.
Okazuje się, że proces umieszczania znaczników aktywnych genów (pozytywna pamięć epigenetyczna) jest katalizowany m.in. przez białko KMT2/MLL, obecne w różnych tkankach. Zaburzenia pracy tego białka wprowadzają zamęt i zmiany "tożsamości" komórek, co w konsekwencji najczęściej wywołuje choroby nowotworowe, ale również psychiczne.
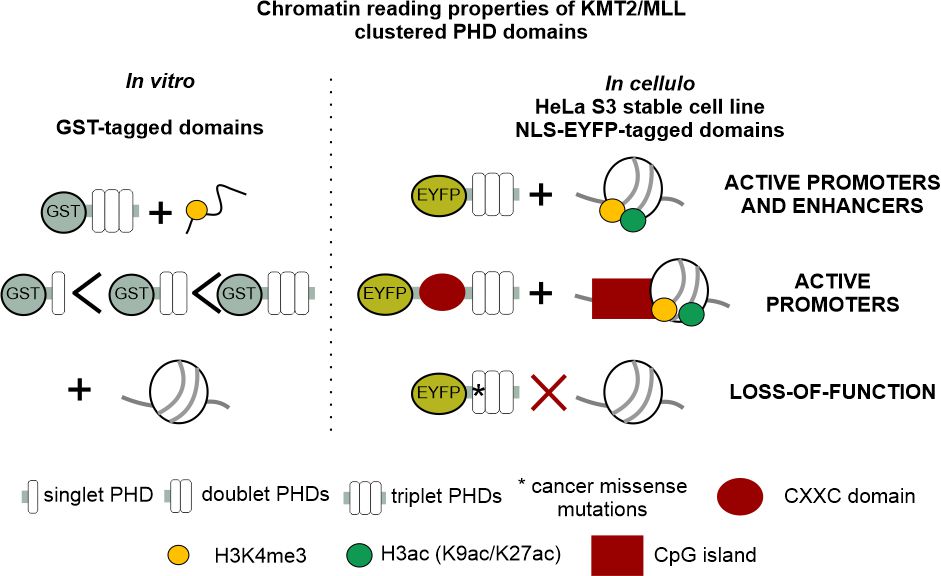
Naukowcy z Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie pod kierownictwem prof. Matthiasa Bochtlera chcieli zrozumieć, w jaki sposób białka KMT2/MLL znajdują swoje cele, aby móc wprowadzić znacznik w obrębie aktywnych genów. W najnowszej pracy Stroynowska-Czerwińska i wsp. pokazują, że małe fragmenty tych białek (tzw. domeny) i interakcje między nimi są wystarczające do tego celu. Autorzy udowadniają również, że nawet pojedyncza, związana z nowotworem, mutacja zmiany sensu w obrębie tych domen może znieść specyficzność ich celowania.
Podsumowując, pamięć genetyczna zależy od pętli sprzężenia zwrotnego, która odpowiada za metylację H3K4, aktywny znacznik chromatyny, na aktywnych promotorach i wzmacniaczach. Do tej pory nie rozumiano, w jaki sposób metylotransferazy KMT2/MLL znajdują swoje cele chromatynowe. Stroynowska-Czerwinska i in. pokazują, że skupione domeny PHD w białkach KMT2/MLL są wystarczające do tego celu. Autorzy wykazali, że znane preferencje promotora lub wzmacniacza poszczególnych metylotransferaz KMT2/MLL można przypisać odpowiednio obecności lub nieobecności w nich domen CXXC. Praca przedstawia pierwszą cało-genomową charakterystykę wiązania domen będacych czytnikami histonów w komórkach. Mutacje genów KMT2/MLL są silnie związane z rakiem. Autorzy pokazują, że nawet pojedyncza związana z rakiem mutacja zmiany sensu w domenach PHD może znieść specyficzność celowania. Dlatego praca ma również implikacje medyczne.”
Artykuł dostępny pod linkiem:
https://link.springer.com/article/10.1007/s00018-022-04651-1
Stroynowska-Czerwinska, A.M., Klimczak, M., Pastor, M. et al. Clustered PHD domains in KMT2/MLL proteins are attracted by H3K4me3 and H3 acetylation-rich active promoters and enhancers. Cell. Mol. Life Sci. 80, 23 (2023). https://doi.org/10.1007/s00018-022-04651-1