Znamy już wyniki konkursu Best Papers Awards 2022! Nagrodzone zostały najlepsze prace naukowe afiliowane w MIBMiK, opublikowane w minionym roku. Do konkursu można było zgłosić każdą pracę eksperymentalną o charakterze naukowym, bez ograniczeń tematycznych. Najlepsze publikacje zostały wybrane przez Jury złożone z Liderów wszystkich Laboratoriów w MIBMiK. Głównym kryterium oceny były treść i znaczenie publikacji, a nie dane bibliometryczne. Liderzy nie mogli głosować na prace z własnego laboratorium. W 2022 roku nagrodzono następujące prace:
- 1 MIEJSCE
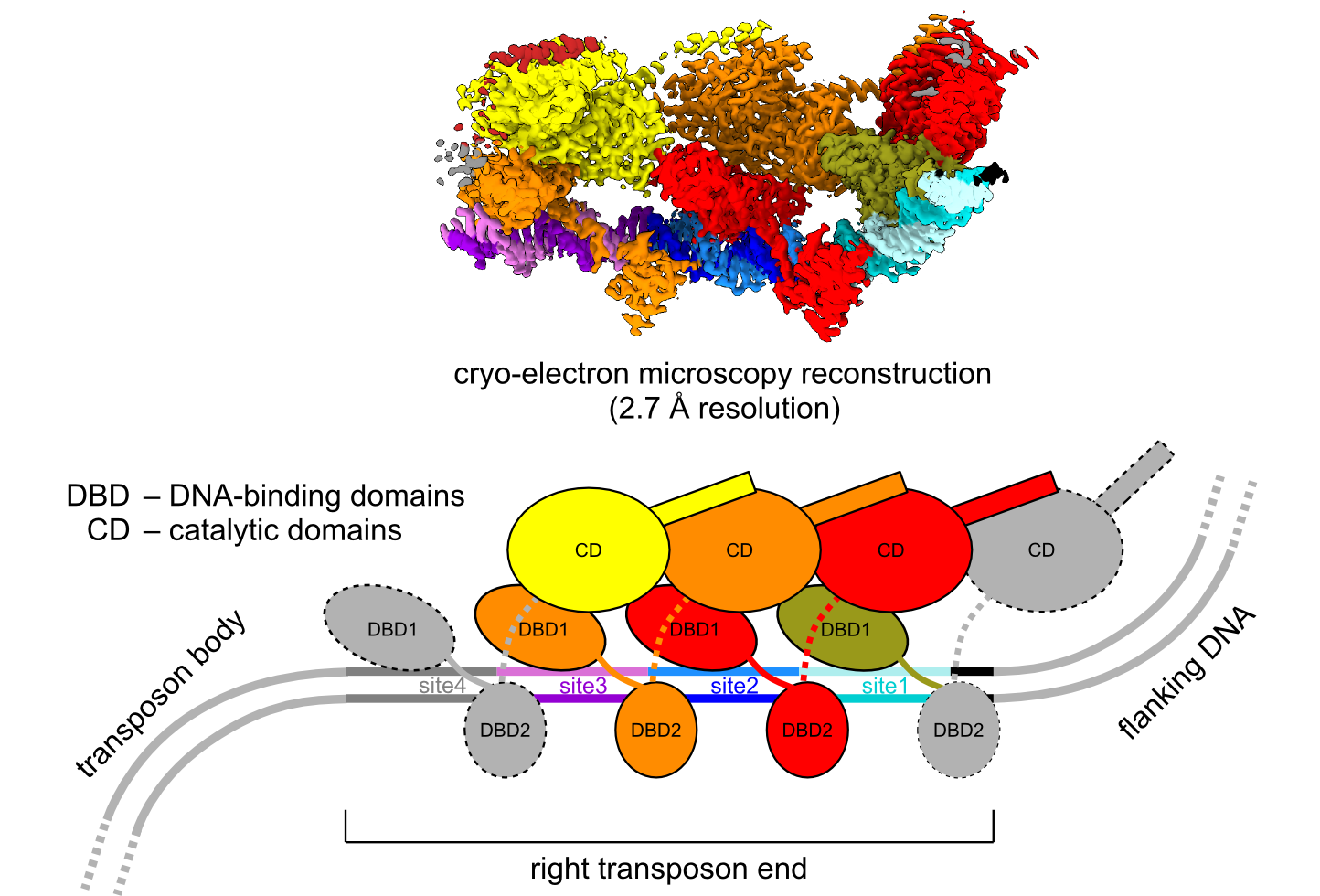
Kaczmarska Z*, Czarnocki-Cieciura M*, Górecka-Minakowska KM*, Wingo RJ, Jackiewicz J, Zajko W, Poznański JT, Rawski M, Grant T, Peters JE#, Nowotny M#. Structural basis of transposon end recognition explains central features of Tn7 transposition systems. Mol Cell, 2022; 82(14):2618-32.e7. doi: 10.1016/j.molcel.2022.05.005
*autorzy z równym wkładem; #autorzy korespondencyjni; pogrubioną czcionką zaznaczono nazwiska autorów z MIBMiK
Transpozony, zwane również „skaczącymi genami”, są fragmentami DNA, które mogą przemieszczać się w obrębie genomów lub pomiędzy nimi w procesie zwanym transpozycją. U bakterii transpozony uczestniczą w przekazywaniu między komórkami genów oporności na antybiotyki i genów wirulencji. Bakteryjny transpozon Tn7 należy do najlepiej zbadanych i najbardziej rozpowszechnionych transpozonów DNA. Przenoszenie Tn7 zachodzi przy udziale pięciu białek kodowanych przez ten element. Tn7 wykorzystuje mechanizm typu "wytnij-wklej", katalizowany przez heteromeryczną transpozazę TnsA-TnsB, która jest rekrutowana do docelowego DNA przez białko TnsC, będące ATPazą typu AAA+. TnsC oddziałuje z jednym z dwóch białek, które kierują transpozon do jego docelowej lokalizacji: TnsD lub TnsE. TnsD kieruje element do zachowanej w ewolucji sekwencji w chromosomie zwanej attTn7, natomiast TnsE umożliwia transpozycję do plazmidów koniugacyjnych. Traspozony związane z CRISPR (ang. CRISPR-associated transposon elements), które wykorzystują systemy CRISPR-Cas do transpozycji DNA kierowanej przez RNA, są spokrewnione z Tn7 i kodują transpozazy podobne do TnsB. Mogą one posłużyć do stworzenia nowych narzędzi do precyzyjnej edycji genów.
Naukowcy z Laboratorium Struktury Białka, kierowanego przez Marcina Nowotnego, we współpracy z grupą Joe Petersa z Cornell University, opisali strukturę i mechanizm działania prototypowej transpozazy TnsB z Tn7 E. coli. Badacze wykorzystali mikroskopię elektronową w reżimie kriogenicznym (cryo-EM) do określenia struktury białka TnsB w kompleksie z dwuniciowym DNA odpowiadającym prawemu końcowi transpozonu o rozdzielczości 2,7 Å. Struktura pokazuje, że kilka łańcuchów białka TnsB, które przyjmuje architekturę domen połączonych elastycznymi łącznikami, oddziałuje z powtarzającymi się miejscami wiązania TnsB w DNA. W wyniku tej interakcji domeny wiążące DNA i domeny katalityczne TnsB układają się naprzemiennie w spleciony sposób. TnsB tworzy niewiele sekwencyjnie specyficznych kontaktów z DNA, co prowadzi do preferencji sekwencyjnej, a nie do ścisłej specyficzności. Szereg cząsteczek TnsB, które wiążą się z wieloma słabo zachowanymi w ewolucji miejscami, które są położone w odpowiednich odstępach, zmienia tę preferencję w specyficzne rozpoznanie końca transpozonu. Naukowcy zaproponowali również model kompleksu białka TnsB z DNA po wycięciu transpozonu i wstawieniu go w nowe miejsce, co pozwala zrozumieć końcowe etapy reakcji prowadzonej przez Tn7 TnsB. Wyniki te pomagają wyjaśnić, w jaki sposób subtelne różnice w odstępach między miejscami wiązania są wykorzystywane do specyficznego rozpoznawania końca transpozonu. Opisane badania wyjaśniają najważniejsze cechy transpozonów Tn7.
- 2 MIEJSCE
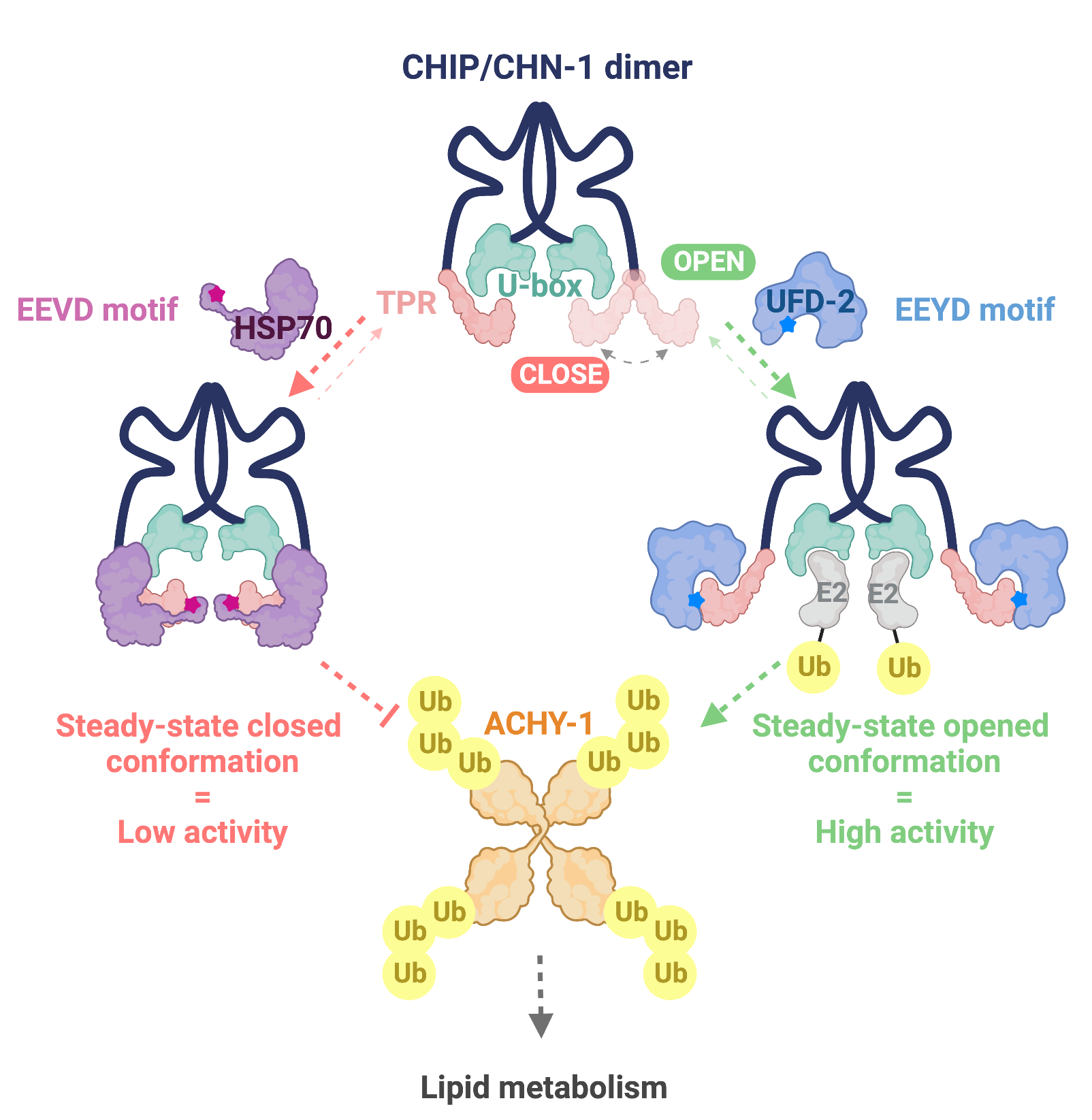
Das A, Thapa P, Santiago U, Shanmugam N, Banasiak K, Dąbrowska K, Nolte H, Szulc NA, Gathungu RM, Cysewski D, Krüger M, Dadlez M, Nowotny M, Camacho CJ, Hoppe T, Pokrzywa W#. A heterotypic assembly mechanism regulates CHIP E3 ligase activity. EMBO J, 2022; 41(15):e109566, doi: 10.15252/embj.2021109566
#autor korespondencyjny; pogrubioną czcionką zaznaczono nazwiska autorów z MIBMiK
Los białek eukariotycznych nadzorowany jest przez sieć białek opiekuńczych (chaperonów) i system ubikwityna-proteasom (UPS). Białko CHIP jest ważną dla kontroli jakości ligazą ubikwityny E3, która łączy system chaperonów z UPS w celu degradacji uszkodzonych białek. Pośredniczy ona również w ubikwitylacji niezależnej od chaperonów i może oddziaływać z innymi ligazami E3. Jednak regulacja działania CHIP i selektywności substratów w odpowiedzi na wiązanie do niego chaperonów i E3 pozostawała niejasna. Naukowcy z Laboratorium Metabolizmu Białek kierowanego przez Wojciecha Pokrzywę przeprowadzili strukturalno-funkcjonalną analizę kompleksu utworzonego przez CHIP i UFD-2, będącą również ligazą ubikwityny E3, kierując się ideą, że stworzą one wydajny system ubikwitylacji stanowiący alternatywę dla osi CHIP/chaperon. Uzyskane dane wykazały, że wiązanie UFD-2 promuje strukturalne wzmocnienie funkcji CHIP. Badacze wykazali również, że białko szoku cieplnego Hsp70 konkuruje z UFD-2 o wiązanie CHIP i negatywnie reguluje aktywność kompleksu poprzez stabilizację CHIP w nieaktywnym stanie. Wykorzystując nicienia Caenorhabditis elegans, naukowcy odkryli, że interakcja z UFD-2 umożliwia CHIP regulację S-adenozylohomocysteinazy, enzymu kluczowego dla metylacji w komórkach. Wyniki uzyskane przez grupę Wojciecha Pokrzywy otwierają nowe horyzonty w badaniach nad współpracą ligaz ubikwityny w uzyskiwaniu wysokiej aktywności i selektywności substratowej. Ponadto, ujawniony mechanizm przełączania procesywności CHIP ma potencjalne zastosowanie w technologii ukierunkowanej degradacji białek.
- 3 MIEJSCE
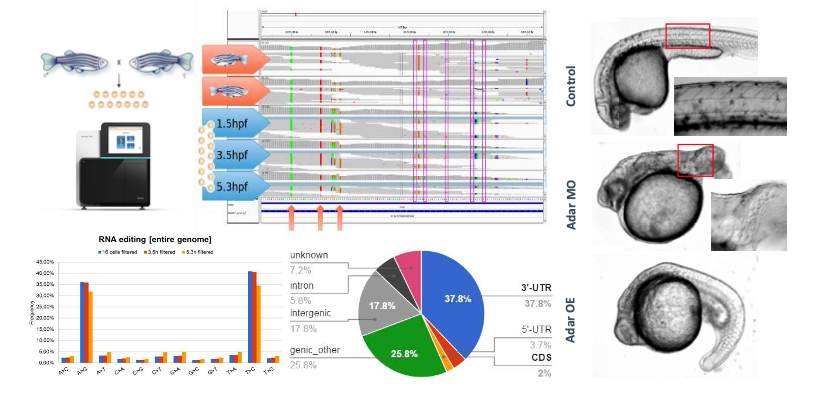
Niescierowicz K*, Pryszcz L*, Navarrete C*, Tralle E*, Sulej A*, Abu Nahia K, Kasprzyk ME, Misztal K, Pateria A, Pakuła A, Bochtler M#, Winata C#. Adar-mediated A-to-I editing is required for embryonic patterning and innate immune response regulation in zebrafish. Nat Commun, 2022; 13(1):5520, doi: 10.1038/s41467-022-33260-6
*autorzy z równym wkładem; #autorzy korespondencyjni; pogrubioną czcionką zaznaczono nazwiska autorów z MIBMiK
Konwersja adenozyny do inozyny (A-do-I) jest niezbędna do regulacji wrodzonego układu odpornościowego u ludzi i innych ssaków oraz jest związana z chorobami człowieka, w tym z chorobami autoimmunologicznymi. Enzym deaminaza adenozyny działająca na RNA (Adar) katalizuje edycję polegającą na deaminacji adenozyny (A) w pozycji C6 i przekształcenie jej w inozynę (I). Naukowcy z Laboratorium Genomiki Rozwoju Danio Pręgowanego i Laboratorium Biologii Strukturalnej, kierowani odpowiednio przez Cecilię Winatę i Matthiasa Bochtlera, badali rolę białka Adar u ryby danio, gdzie ulega ono wysokiej ekspresji w najwcześniejszych etapach embriogenezy. Śledzenie edycji RNA w całym genomie ryby poprzez połączone analizy genomu rodzicielskiego i transkryptomu embrionalnego ujawniła powszechną edycję A-do-I w matczynych i najwcześniejszych zygotycznych transkryptach, z których większość wystąpiła w rejonie 3' niepodlegającym translacji. Transkrypty, o których wiadomo, że odgrywają rolę w gastrulacji i kształtowaniu zarodka, zawierały wiele miejsc edycji, co sugeruje, że Adar może wywierać swoją funkcję za ich pośrednictwem. Eksperymentując z utratą i wzmocnieniem funkcji tego białka, badacze wykazali, że matczyny Adar z nienaruszoną domeną deaminazy jest konieczny do prawidłowego kształtowania się zarodka wzdłuż osi pionowej i strzałkowej. Analiza zygotycznych mutantów ujawniła odrębną funkcję białka Adar pochodzącego z zygoty w regulacji wrodzonej odpowiedzi immunologicznej, która to rola jest zachowana u ssaków. Podsumowując, badania wykazały udział edycji A-do-I przez Adar w kształtowaniu się zarodka i pokazały ewolucyjnie konserwowane znaczenie tego białka u ryb i ssaków.