Naukowcy zbadali jak bakterie bronią się przed wirusami
Badacze z Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie, wspólnie z naukowcami z Wydziału Eksperymentalnych Nauk Medycznych Uniwersytetu w Lund przeprowadzili szczegółowe badanie białka AbiA - enzymu zaangażowanego w obronę bakterii przed infekcjami wirusowymi. Opisane w Nucleic Acid Research analizy strukturalne i biochemiczne białek, uzupełnione o badania in vivo, rzucają światło na wcześniej niezbadany mechanizm działania AbiA.
Bakterie, podobnie jak wszystkie organizmy żywe, są narażone na infekcje wirusowe. Aby im zapobiec lub nie dopuścić do replikacji wirusów po zarażeniu, drobnoustroje stosują różne strategie obronne. Wiele białek bakteryjnych zaangażowanych w obronę przeciwwirusową jest klasyfikowanych jako odwrotne transkryptazy (RT). Typowe RT wykorzystują RNA jako matrycę – wzorzec, na podstawie którego tworzą kopie informacji genetycznej w postaci DNA. Przewiduje się, że pewne bakteryjne RT odpowiadają za zaprogramowaną śmierci komórki, określaną jako infekcja abortywna. Zaatakowana bakteria popełnia więc „samobójstwo”, aby aby nie pozwolić na namnażanie wirusa.
W opisywanym badaniu zespół Laboratorium Struktury Białka MIBMiK pod kierownictwem Marcina Nowotnego określił pierwsze atomowe struktury przestrzenne jednej z bakteryjnych RT – białka AbiA, ze związanym DNA produkowanym przez ten enzym. Badania ujawniły istotne różnice między AbiA a innymi transkryptazami typu Abi. AbiA tworzy dimer (połączenie dwóch jednakowych cząsteczek). Oprócz domeny podobnej do RT, która jest odpowiedzialna za produkcję DNA, i domeny złożonej z α-helis, która stabilizuje produkowane DNA, AbiA zawiera też domenę HEPN wspólną dla wielu białek przeciwwirusowych. Eksperymenty in vitro, w których badana była aktywność enzymu, wykazały, że do zainicjowania syntezy DNA AbiA wykorzystuje mechanizm primingu białkowego (pierwszy nukleotyd produkowanej nici DNA przyczepiany jest do jednej z reszt białka, a nie, jak w większości przypadków, do istniejącego już fragmentu kwasu nukleinowego) i może wytwarzać fragmenty DNA o średniej wielkości około 100 nukleotydów. Analiza sekwencji tych produktów DNA przeprowadzona przez badaczy z Laboratorium Biologii RNA wykazała, że enzym ma silną preferencję dla adenozyny i cytydyny (a więc dwóch z czterech podstawowych nukleotydów wchodzących w skład DNA), w przeciwieństwie do enzymu AbiK, który wytwarza znacznie dłuższe produkty o całkowicie losowej sekwencji. Ponadto przeprowadzone przez zespół współpracowników z Uniwersytetu w Lund testy infekcji wirusowej, wykazały, że do antyfagowego działania AbiA wymagana jest aktywność polimerazy DNA. Wyniki te sugerują ponadto, że wbrew dotychczasowym założeniom, mechanizm obronny AbiA nie polega na indukowaniu samobójstwa zainfekowanej komórki.
Z artykułem “Structure-functional characterization of Lactococcus AbiA phage defense system” można zapoznać się pod linkiem: https://academic.oup.com/nar/article/52/8/4723/7642066?searchresult=1
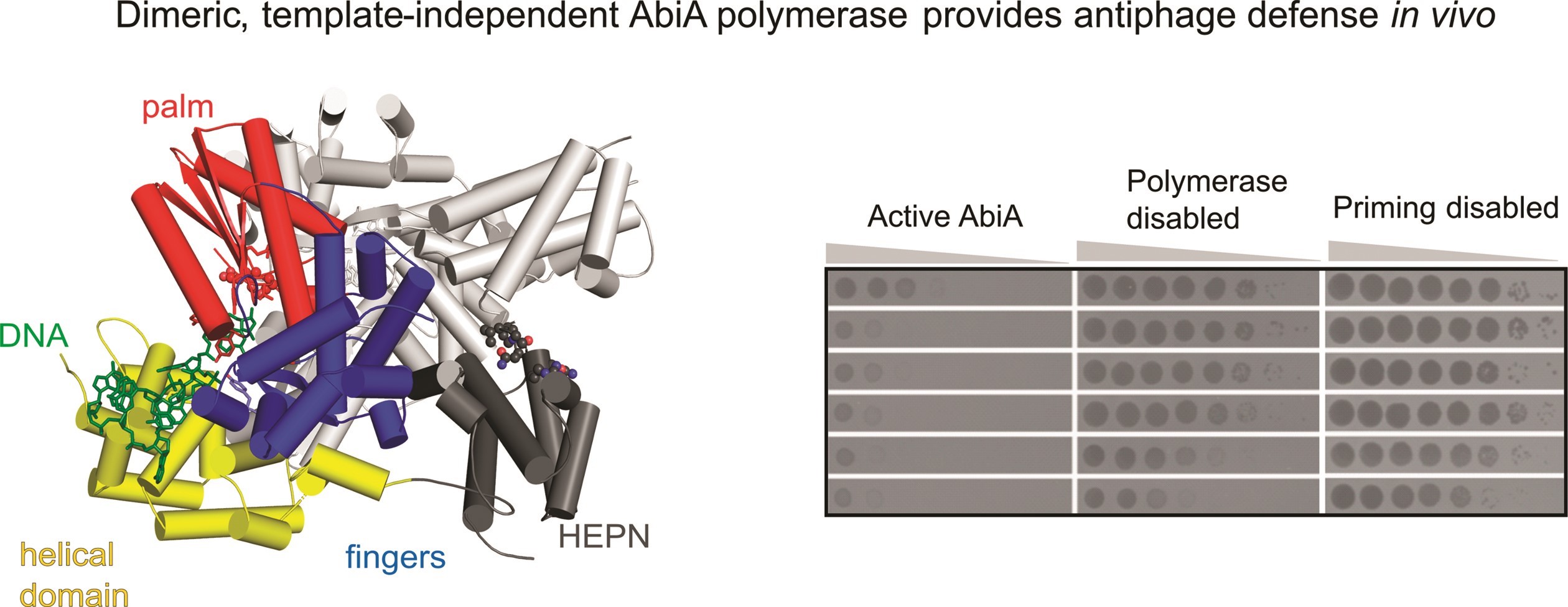 Ogólna struktura kompleksu L. lactis AbiA-DNA (po lewej) i test aktywności antyfagowej AbiA typu dzikiego oraz jego zmutowanych wariantów (po prawej).
Ogólna struktura kompleksu L. lactis AbiA-DNA (po lewej) i test aktywności antyfagowej AbiA typu dzikiego oraz jego zmutowanych wariantów (po prawej).
