Kluczowym przesłaniem artykułu jest odkrycie u danio pręgowanego zjawiska edycji RNA od A do I, polegającego na modyfikacji kodu genetycznego RNA w stosunku do kodu zakodowanego w DNA. Stwierdzono również, że proces ten jest niezbędny do prawidłowego rozwoju embrionalnego we wczesnym okresie życia zabrafish, natomiast w późniejszym jego okresie jest wymagany do regulacji wrodzonej odpowiedzi immunologicznej.
Edycja A do I jest niezbędna do regulacji wrodzonego układu odpornościowego u ludzi i ssaków oraz ma związek z chorobami człowieka, w tym schorzeniami autoimmunologicznymi – tak wynika z badania przeprowadzonego przez grupę badaczy, m.in. dr hab. Cecilie Winata oraz prof. Matthiasa Bochtlera z Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie. Odkrycie konserwatywnej roli u zebrafish otwiera nowe możliwości modelowania pokrewnych chorób ludzkich w tym organizmie modelowym. Dzięki temu możliwe będzie lepsze zrozumienie mechanizmu choroby i informowanie o przyszłym rozwoju terapii. Ponadto, precyzyjna edycja kwasu nukleinowego przy użyciu enzymów edytujących RNA, takich jak ADAR, może być potencjalnie wykorzystana do rozwoju ukierunkowanej terapii genetycznej.
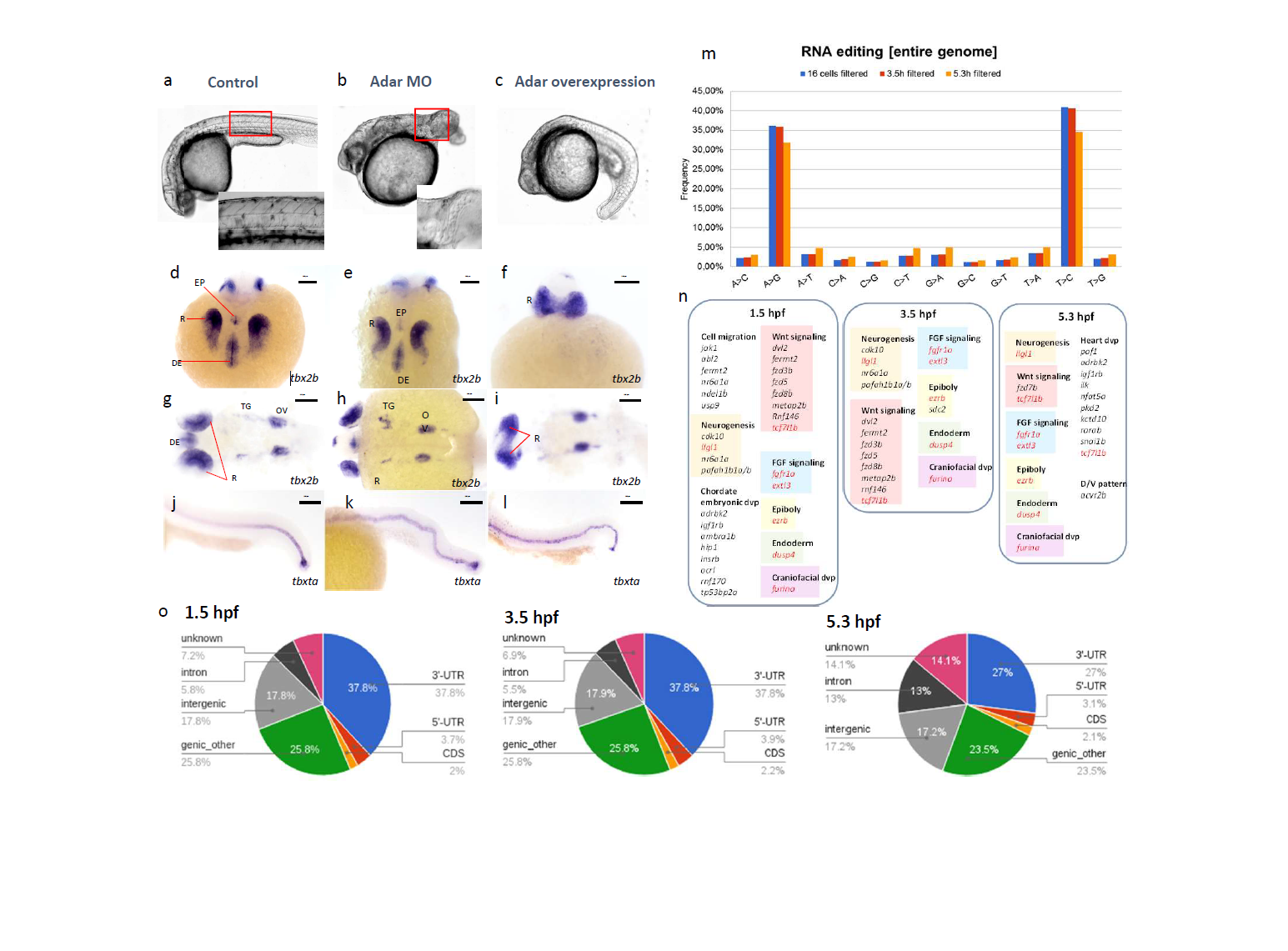
W tej publikacji autorzy opisują różne matczyne i zygotyczne funkcje edycji RNA z udziałem ADAR w kształtowaniu embrionalnym wzdłuż osi przednio-tylnej i tylno-dolnej oraz w regulacji wrodzonej odpowiedzi immunologicznej.W tym manuskrypcie autorzy opisują różne matczyne i zygotyczne funkcje edycji RNA z udziałem ADAR w kształtowaniu embrionalnym wzdłuż osi przednio-tylnej i tylno-dolnej oraz w regulacji wrodzonej odpowiedzi immunologicznej.Redagowanie A do I polega na deaminacji adenozyny (A) w pozycji C6, co prowadzi do powstania inozyny (I). Za katalizowanie tego procesu odpowiedzialne są enzymy znane jako ADAR (Adenosine Deaminase Acting on RNA). Adar u Zebrafish ulega wysokiej ekspresji we wczesnych etapach embriogenezy. Poprzez eksperymenty z utratą lub wzmocnieniem funkcji Adar wykazaliśmy, że matczyna funkcja Adar jest niezbędna do ustanowienia przednio-tylnej i dorso-centralnej osi ciała, jak również do kształtowania embrionalnego wzdłuż tych osi. Funkcja ta jest zależna od nienaruszonej domeny deaminazy. Odkrycie edycji w całym genomie poprzez połączone analizy genomu rodzicielskiego i transkryptomu embrionalnego ujawniło powszechną edycję A do I w matczynych i najwcześniejszych zygotycznych transkryptach, z których większość wystąpiła w regionie 3'-UTR. Transkrypty znane z roli w gastrulacji i kształtowaniu embrionalnym zawierały wiele miejsc edycji, co sugeruje, że Adar może wywierać swoją funkcję za ich pośrednictwem. Redukcja lub nadekspresja Adaru wpłynęła na globalne wzorce edycji i profil transkryptomu obserwowalny do 12 hpf, co zbiega się z czasem, kiedy zaczynają się pojawiać najwcześniejsze wzorce embrionalne. Wreszcie, analiza zygotycznych mutantów Adar ujawniła odrębną zygotyczną funkcję Adar w regulacji wrodzonej odpowiedzi immunologicznej, która to rola jest zachowana w stosunku do ssaków. Podsumowując, nasze badania wykazały nową funkcję Adaru w regulacji kształtowania zarodka i ujawniły zachowanie zygotycznej funkcji Adaru między zebrami i ssakami. Rycina 1. (A-C) Defekty fenotypowe w 24 hpf spowodowane przez knockdown i nadekspresję Adar. Zarodki z nadekspresją Adar rozwijają nieprawidłowy fenotyp w tylnej części z zaburzoną osią ciała, skróconym ogonem i krzywym, zdezorganizowanym notochordem. Fenotyp MO może być w pełni uratowany przez wstrzyknięcie mRNA typu dzikiego. (D-I) Ekspresja tbx2b zaznaczająca diencephalon (DE), R, epiphysis (EP), trigeminal ganglion (TG) i otic vesicle (OV) u Adar morpholino knockdown i adar mRNA overexpression. (J-L) Ekspresja tbxta zaznacza notochord. (M) Niedopasowania między danymi sekwencjonowania RNA i DNA. Ponieważ biblioteki RNA nie były selektywne, niedopasowania były odczytywane jako ich uzupełnienie (tj. T->C zamiast A->G lub C->T jako G->A) w około połowie przypadków. (N) Wybrane transkrypty genów ścieżek sygnałowych rozwoju, zaangażowanych w rozwój dorsoventralny i/lub anteroposterioralny, zawierające dwa lub więcej miejsc edycji RNA w ich regionie 3'-UTR. Nazwy genów w kolorze czerwonym oznaczają te, które są powszechnie edytowane na wszystkich trzech etapach. (O) Związek miejsc edycji z cechami genomowymi. Duża część edycji RNA jest klasyfikowana jako `genic_other` z powodu nakładania się intronów/eksonów/UTR z wielu transkryptów.