News
W dniach 6-7 czerwca 2023 r. reprezentacja MIBMiK składająca się z przedstawicieli dyrekcji, naukowców i administracji aktywnie uczestniczyła w konferencji Envisioning the research centres of the future zorganizowanej z okazji 10. rocznicy powstania EU-LIFE w Lizbonie w Portugalii.
Konferencja stworzyła żywą i interaktywną przestrzeń do wymiany pomysłów i dyskusji na temat tego, jak być lepszymi badaczami, bardziej kreatywnymi, proaktywnymi, odpowiedzialnymi i angażującymi. Zgromadziła społeczność naukową, w tym badaczy i liderów naukowych, administratorów nauki i decydentów politycznych, aby wspólnie zastanawiać się nad idealnym miejscem badawczym przyszłości.
Podczas dyskusji panelowych prof. Andrzej Dziembowski, kierownik Laboratorium Biologii RNA – Grupa ERA Chairs, mówił o roli kreatywności w nauce, prof. Janusz Bujnicki, kierownik Laboratorium Bioinformatyki i Inżynierii Białka, zaś o tym, jak być produktywnym w świecie nauki. Dr Małgorzata Figiel przedstawiła słuchaczom wnioski z dyskusji na temat kreatywności. MIBMiK zaprezentował również dwa plakaty: "Centre of Excellence in RNA and Cell Biology - IIMCB of the future", którego współautorami są dr Iwona Pilecka, Dorota Libiszowska, Marcin Ogonowski i Alexia Danyłow oraz "Scientific excellence has no gender: IIMCB’s activities on gender equality, diversity & inclusion", którego współautorkami są Agnieszka Faliszewska, Katarzyna Fiedorowicz i dr Urszula-Białek Wyrzykowska.
Konferencję poprzedziło spotkanie społeczności EU-LIFE. Odbyło się w Instituto Gulbenkian de Ciência w Oeiras, gdzie przedstawiciele instytutów zreszonych w ramach EU-LIFE omawiali tematy przewodnie w ramach grupach roboczych.
EU-LIFE to konsorcjum niezależnych instytutów badawczych, którego misją jest wspieranie i wzmacnianie europejskiej doskonałości naukowej. Organizację tworzą cieszące się międzynarodowym uznaniem instytucje, które prowadzą wysokiej jakości badania i aktywnie dzielą się wiedzą i wspierają rozwój talentów. Od momentu założenia w 2013 r., EU-LIFE stało się interesariuszem w rozwoju polityki naukowej regularnie uczestnicząc w dialogu na temat europejskiej polityki naukowej. Od 2000 r. MIBMiK jest jedyną polską instytucją zrzeszoną w EU-LIFE z 14 innymi instytutami w celu osiągnięcia i utrzymania doskonałości w naukach o życiu, kładąc nacisk na jakość i odpowiedzialną naukę oraz podkreślając kwestie związane z europejską polityką naukową.
Międzynarodowy Instytut Biologii Molekularnej i Komórkowej w Warszawie ogłasza nabór na stanowiska Menedżera ds. Własności Intelektualnej i Menedżera ds. Transferu Technologii. Zapraszamy do zapoznania się z ofertą.
Intellectual Property Manager
The International Institute of Molecular and Cell Biology in Warsaw (IIMCB), Poland
invites applications for the position of Intellectual Property Manager
Are you passionate about knowledge valorization and are you ready to make a meaningful impact by translating IIMCB research discoveries into real-world applications? Are you open to expanding your expertise in intellectual property interests by engaging in a training-in-residence at the Flanders Institute for Biotechnology (VIB) in Belgium, our strategic partner? Are you ready to work in a dynamic team taking on the challenges of IP protection and technology transfer on an international level? If yes, please apply.
The IIMCB hosts a vibrant, multinational community of scientists and is well connected internationally as exemplified by the composition of the IIMCB's International Advisory Board and the IIMCB's membership in EU-LIFE, an alliance of 15 top European research institutions. The IIMCB is on its way to unprecedented scientific and institutional growth. Through expanding by 2025 to 20 research groups involved in innovative translational projects in RNA biology and cell biology, we aim to become a unique Polish center where excellent science is combined with professional IPR protection and commercialization. To this end, the IIMCB is establishing institutional support for translating basic science discoveries and research services into commercial applications and clinical innovations. The Intellectual Property Manager will be one of the key people in these processes. The successful candidate will focus on managing the intellectual property assets of the IIMCB by providing professional support in the identification, drafting of patent applications and licensing of IIMCB inventions. They will become a core staff of a dedicated technology transfer unit at the IIMCB and will benefit from a one-year training-in-residence at the VIB located in Ghent, Belgium, where they will gain expertise in identifying and processing intellectual property.
The establishment of a comprehensive system of technology transfer support is a part of the IIMCB’s institutional project entitled “RNA and Cell Biology - from Fundamental Research to Therapies” (RACE) funded in the Teaming for Excellence programme under Horizon Europe.
Key responsibilities:
- Creating an environment at the IIMCB that fosters innovation and entrepreneurial thinking
- Engaging with researchers to inspire and motivate them to think about the relevance of their research to society and the practical application of their discoveries
- Monitoring the results of research conducted in the IIMCB laboratories to determine their commercialization potential and recommending whether intellectual property protection should be sought
- Carrying out analyses of costs associated with patent prosecution
- Conducting patenting procedures (searching, drafting and prosecution)
- Organizing, managing and controlling all patents, trademarks and copyright files
- Overseeing IP related issues in externally funded research projects, including drafting IP agreements and managing IP during projects implementation
- Providing IP training to IIMCB researchers and advice on all IP-related matters
Necessary qualifications:
- PhD in life sciences
- Experience in biomedical research
- Hands-on knowledge of patent procedures is an asset
- Analytical mind, capable of analyzing a broad spectrum of life science topics and with the ability to deep-dive into scientific content
- Readiness for a 1-year training-in-residence at VIB, Ghent, Belgium
- High multitasking capacity combined with the ability to respect deadlines
- Excellent interpersonal and communication skills
- Fluency in oral and written Polish and English
The IIMCB offers:
- Employment contract with the IIMCB and secondment for 1-year training-in-residence at VIB, Ghent, Belgium
- Full-time salary in the range of 9. 000-11. 000 PLN gross, including the period of secondment, and an additional allowance for a secondment of 10. 400 PLN gross
- Full-time salary is complemented by an additional 13th salary* plus an annual bonus.
- Attractive social package (co-financing Multisport cards, Christmas, Easter and summer holiday bonuses, etc.)
- Additional paid vacation time starting from the 2nd year of employment
- Work in hybrid mode
- Professional development and business travel opportunities
- Full organizational and administrative support by professional English-speaking staff
- Friendly, inclusive, international working environment
The application should include:
- Cover letter explaining the motivation for joining the IIMCB
- Curriculum Vitae
- A concise statement of 1-3 professional achievements
- Contact information for two references
Applications should be sent to Ten adres pocztowy jest chroniony przed spamowaniem. Aby go zobaczyć, konieczne jest włączenie w przeglądarce obsługi JavaScript., in the subject please include "IPM" and your first and last name.
The deadline for applications is June 15, 2023.
Job interviews are scheduled for June 22-23, 2023.
Applications should include the statement: “I hereby agree to the processing of my personal data, included in the application documents by the International Institute of Molecular and Cell Biology in Warsaw, 4 Księcia Trojdena Street, 02-109 Warsaw, for the purpose of carrying out the current recruitment process.” Your personal data will be processed only for the purpose of the recruitment procedure by the International Institute of Molecular and Cell Biology in Warsaw. Full information is available at https://bit.ly/3UFWpY2.
*13th salary is calculated as 8.5% of the gross salary received in a given calendar year (excluding sick pay) under the condition of having worked a minimum of 6 months in a given year.
Technology Transfer Manager
The International Institute of Molecular and Cell Biology in Warsaw (IIMCB), Poland
invites applications for the position of Technology Transfer Manager
Are you passionate about knowledge valorization and are you ready to make a meaningful impact by translating IIMCB research discoveries into real-world applications? Are you open to expanding your expertise by engaging in the training-in-residence at the Flanders Institute for Biotechnology (VIB) in Belgium, our strategic partner? Are you eager to build a dynamic team taking on the challenges of IP protection and technology transfer on an international level? If yes, please apply.
The IIMCB hosts a vibrant, multinational community of scientists and is well connected internationally as exemplified by the composition of the IIMCB's International Advisory Board and the IIMCB's membership in EU-LIFE, an alliance of 15 top European research institutions. The IIMCB is on its way to unprecedented scientific and institutional growth. By expanding to 20 research groups involved in innovative translational projects in RNA biology and cell biology by 2025, we aim to become a unique Polish center where excellent science is combined with professional IPR protection and commercialization. To this end, the IIMCB is establishing institutional support for translating basic science discoveries and research services into commercial applications and clinical innovations. The Technology Transfer Manager will be at the heart of these processes acting as an interface between the IIMCB scientists and potential clients, including clinics and businesses. They will become a core staff of the dedicated technology transfer unit at the IIMCB and will develop an array of activities for effective IIMCB knowledge valorization. The Technology Transfer Manager will benefit from 1.5-year training-in-residence at the Flanders Institute for Biotechnology (VIB) located in Ghent, Belgium where they will gain expertise in identifying intellectual property with commercialization potential, validating research discoveries, establishing partnerships with industry, and using various knowledge transfer models.
The establishment of a comprehensive system of technology transfer support is a part of the IIMCB’s institutional project entitled “RNA and Cell Biology - from Fundamental Research to Therapies” (RACE) funded in the Teaming for Excellence programme under Horizon Europe.
Key responsibilities:
- Creating an environment at the IIMCB that fosters innovation and entrepreneurial thinking
- Engaging with researchers to inspire and motivate them to think about the relevance of their research to society and the practical application of their discoveries
- Monitoring the results of research conducted in the IIMCB laboratories and ensuring proper protection of results with commercialization potential
- Seeking funding sources and business partners for the development of inventions
- Applying for grants for technology development and commercialization, implementing and reporting on them
- Building and presenting business cases and marketing materials with the aim of partnering with the biotech industry (R&D collaborations and/or licensing)
- Expanding the IIMCB's business network and building the IIMCB's image as a business partner
- Cooperating with lawyers and patent attorneys in preparation of legal documents and their follow up (alliance management)
- Monitoring IIMCB discoveries along the patenting and commercialization processes
Necessary qualifications:
- PhD in life sciences
- Experience in biomedical research
- Experience in translational projects and technology transfer is an asset
- Strong interest to develop professional competences in technology transfer
- Readiness for a 1.5-year training-in-residence at VIB, Ghent, Belgium
- Strong analytical skills
- Decision-making and negotiation skills
- Excellent interpersonal and communication skills
- Fluency in oral and written Polish and English
The IIMCB offers:
- Employment contract with the IIMCB and secondment for 1.5-year training-in-residence at VIB, Ghent, Belgium
- Full-time salary in the range of 12.000-14.000 PLN gross, including the period of secondment, and an additional allowance for secondment of 10.400 PLN gross
- Full-time salary is complemented by an additional 13th salary* plus an annual bonus
- Attractive social package (co-financing Multisport cards, Christmas, Easter and summer holiday bonuses, etc.)
- Additional paid vacation starting from the 2nd year of employment
- Work in hybrid mode
- Professional development and business travel opportunities
- Full organizational and administrative support by professional English-speaking staff
- Friendly, inclusive, international working environment
The application should include:
- Cover letter explaining the motivation for joining the IIMCB
- Curriculum Vitae
- A concise statement of 1-3 professional achievements
- Contact information for two references
Applications should be sent to Ten adres pocztowy jest chroniony przed spamowaniem. Aby go zobaczyć, konieczne jest włączenie w przeglądarce obsługi JavaScript., in the subject please include "TTM" and your first and last name.
The deadline for applications is June 15, 2023.
Job interviews are scheduled for June 22-23, 2023.
Applications should include the statement: “I hereby agree to the processing of my personal data, included in the application documents by the International Institute of Molecular and Cell Biology in Warsaw, 4 Księcia Trojdena Street, 02-109 Warsaw, for the purpose of carrying out the current recruitment process.” Your personal data will be processed only for the purpose of the recruitment procedure by the International Institute of Molecular and Cell Biology in Warsaw. Full information is available at https://bit.ly/3UFWpY2.
*13th salary is calculated as 8.5% of the gross salary received in a given calendar year (excluding sick pay) under the condition of having worked a minimum of 6 months in a given year.
Dr Aleksandra Anna Kołodziejczyk z nowym grantem NCN!
Dr Aleksandra Kołodziejczyk, kierowniczka Laboratorium Genomiki Komórkowej w Międzynarodowym Instytucie Biologii Molekularnej i Komórkowej w Warszawie, otrzymała finansowanie w ramach konkursu SONATA 18 z funduszy Narodowego Centrum Nauki (NCN). Grant w wysokości 2 544 840 PLN przeznaczony jest na projekt zatytułowany: "Rola osi jelito-wątroba w zatruciu grzybami z gatunku Amanita". Dr Kołodziejczyk zajęła pierwsze miejsce na liście rankingowej nagrodzonych projektów w ramach panelu NZ5. Serdecznie gratulujemy!
Celem projektu jest kompleksowe zrozumienie mechanizmów molekularnych i komórkowych leżących u podstaw patofizjologii zatrucia toksynami muchomora sromotnikowego z myślą o poprawie wyników klinicznych. Opis projektu dostępny jest tutaj.
Projekt potrwa 36 miesięcy i będzie realizowany przez konsorcjum dwóch podmiotów: Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie oraz Warszawskiego Uniwersytetu Medycznego.
Dr hab. Cecilia Winata – laureatką konkursu OPUS 24!
Dr hab. Cecilia Winata, kierowniczka Laboratorium Genomiki Rozwoju Danio Pręgowanego w Międzynarodowym Instytucie Biologii Molekularnej i Komórkowej w Warszawie, otrzymała finansowanie w ramach konkursu OPUS 24 z funduszy Narodowego Centrum Nauki (NCN). Grant w wysokości 3 040 240 PLN przeznaczony jest na projekt zatytułowany: "Wyjaśnianie wkładu niekodujących elementów genomowych w rozwój i choroby serca w skali pojedynczych komórek ". Dr hab. Cecilia Winata zajęła pierwsze miejsce na liście rankingowej nagrodzonych projektów w ramach panelu NZ2. Serdecznie gratulujemy!
Projekt potrwa 48 miesięcy. Prowadzone badania mają na celu odkrycie nowych czynników i mechanizmów genetycznych powodujących wrodzoną chorobę serca i wnieść krytyczny wgląd w zrozumienie mechanizmu niekodujących wariantów genetycznych w patogenezie tej choroby. Opis projektu dostępny jest tutaj.
W dniu 11 maja 2023 r. prof. Marta Miączyńska, Dyrektor Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie podpisała umowę na przygotowanie dokumentacji projektowej nowej siedziby Instytutu z pracownią architektoniczną Atelier Tektura Sp. z o. o, zwycięzcą w konkursie na najlepszą koncepcję architektoniczną siedziby Instytutu.
„Podpisanie umowy to kolejny krok przybliżający nas do nowej siedziby, tak ważnej dla dalszego rozwoju Instytutu. Jesteśmy przekonani, iż współpraca z Atelier Tektura będzie przebiegała pomyślnie i pozwoli w zaplanowanym czasie zrealizować postawiony cel” – powiedziała prof. Marta Miączyńska, Dyrektor MIBMiK
W grudniu 2022 r. został rozstrzygnięty konkurs na koncepcję architektoniczną nowej siedziby Instytutu, którego zwycięzcą została Atelier Tektura. Nagrodzony projekt został doceniony za bardzo dobre, racjonalne oraz logiczne rozwiązanie rozkładu pomieszczeń. Doceniono również elastyczność projektu, czyli możliwość modyfikacji rozkładu jak i rozwiązań konstrukcyjnych.
4 dyscypliny naukowe, 9 renomowanych Instytutów badawczych, 1 wyjątkowy, interdyscyplinarny program kształcenia – tak prezentuje się marka Warszawskiej Szkoły Doktorskiej Nauk Ścisłych i Biomedycznych Warsaw4Phd. Jej interdyscyplinarny program doktorancki oferuje najnowocześniejsze możliwości badawcze w obszarach fizyki, chemii, biologii i medycyny, a także współpracę z wiodącymi ośrodkami naukowymi na całym świecie. Dokładnie 4 maja br. rozpoczęła się międzynarodowa kampania promująca Szkołę Doktorską w mediach społecznościowych. Jej celem jest zaprezentowanie pełnej oferty Szkoły i zachęcenie potencjalnych doktorantów do studiowania w Warszawie. Kampania zadebiutowała filmem promocyjnym, który można obejrzeć TUTAJ. Już 22 maja startuje pierwsza w tym roku rekrutacja do Szkoły Doktorskiej; o szczegółach tego procesu i projektach badawczych, do których można aplikować, piszemy w tekście poniżej.
Nauka staje się coraz bardziej interdyscyplinarna. Aby odkryć coś naprawdę nowego i przełomowego – trzeba stosować różne metody badawcze. Tylko synergia różnych dyscyplin naukowych może prowadzić do fascynujących odkryć. Właśnie taką ofertę ma Szkoła Doktorska Warsaw4PhD, w której obecnie studia doktoranckie realizują studenci z 25 różnych krajów. Dzięki stałej i długotrwałej współpracy z zagranicznymi ośrodkami badawczymi doktoranci mają duże możliwości rozwojowe, również w ramach współpracy międzynarodowej. Uzyskanie doktoratu otwiera możliwość kontynuowania kariery naukowej w najbardziej nowoczesnych laboratoriach na całym świecie.
Rekrutacja w ramach najbliższego naboru rozpocznie się w dniu 22 maja br. i potrwa do 4 czerwca. Projekty badawcze, w ramach których których potencjalni doktoranci mogą przesyłać swoje aplikacje sa opisane TUTAJ.
Warszawską Szkołę Doktorską nauk Ścisłych i Biomedycznych tworzą:
Instytut Biologii Doświadczalnej Polskiej Akademii Nauk
Instytut Chemii Organicznej Polskiej Akademii Nauk
Instytut Chemii Fizycznej Polskiej Akademii Nauk
Instytut Fizyki Polskiej Akademii Nauk
Centrum Fizyki Teoretycznej Polskiej Akademii Nauk
Instytut Wysokich Ciśnień Polskiej Akademii Nauk
Narodowy Instytut Onkologii im. Marii Skłodowskiej-Curie, Państwowy Instytut Badawczy
Instytut Psychiatrii i Neurologii
Międzynarodowy Instytut Biologii Molekularnej i Komórkowej w Warszawie
Więcej szczegółów: https://warsaw4phd.eu/
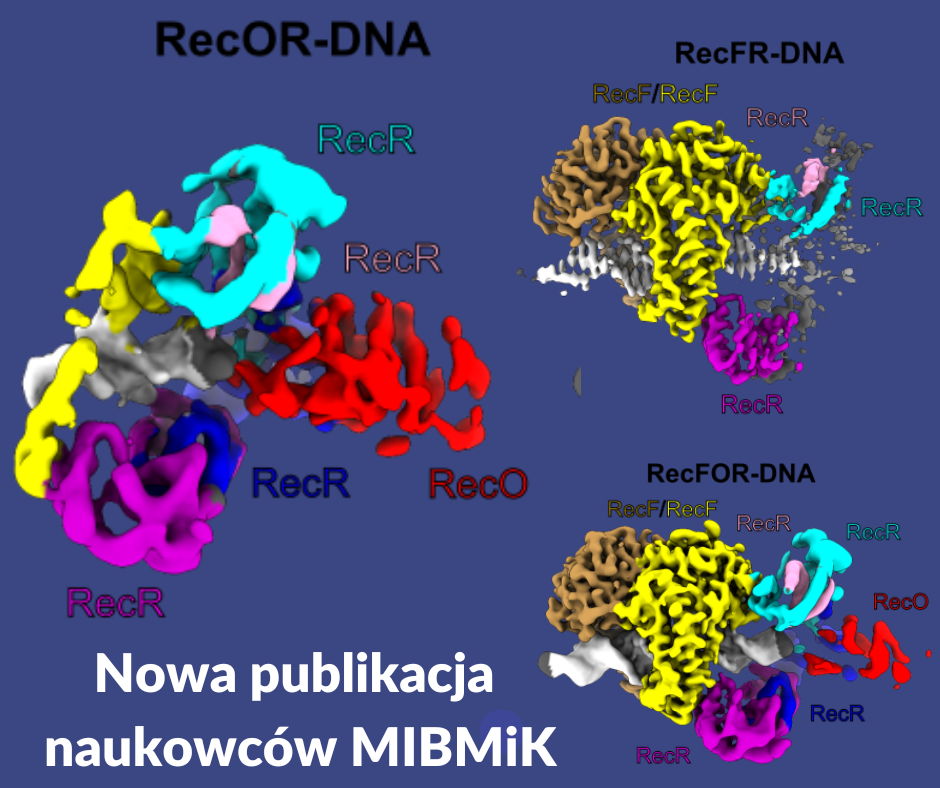
Naukowcy z Laboratorium Struktury Białka Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie wyjaśnili mechanizm działania kluczowego kompleksu białkowego w bakteryjnej rekombinacji homologicznej.
Rekombinacja homologiczna jest jedną z podstawowych ścieżek naprawy uszkodzonej informacji genetycznej. Proces ten polega ona na wytworzeniu jednoniciowej cząsteczki DNA w miejscu uszkodzenia. Następnie, fragment ten jest wykorzystany do poszukiwania matrycy w oparciu o homologię. W przypadku bakterii w jedną ze ścieżek naprawy DNA opartych na rekombinacji homologicznej zaangażowane są białka RecF, RecO i RecR. Białka te wiążą się na styku jednoniciowego (ss) i dwuniciowego (ds) DNA, a następnie ułatwiają wymianę białka SSB (pokrywa ono początkowo ssDNA) na białko RecA, które promuje poszukiwanie sekwencji homologicznej. Dotychczas molekularny mechanizm działania białek RecFOR w tym szlaku był w dużej mierze nieznany. Aby wyjaśnić mechanizm współpracy tych białek w rozpoznawaniu połączeń ss-dsDNA, Shivlee Nirwal i współpracownicy z Laboratorium Struktury Białka, pod kierownictwem prof. Marcina Nowotnego, zbadali struktury kompleksów RecF-DNA i RecFOR-DNA oraz ich subkompleksów. Użyli do tego najnowocześniejszych technik kriomikroskopii elektronowej, korzystając z mikroskopów w Narodowym Centrum Promieniowania Synchrotronowego SOLARIS.
Struktury subkompleksów RecF-DNA i RecFR-DNA pokazują, jak dimer białka RecF wykorzystuje helikalne wypustki do wiązania dwuniciowej cząsteczki DNA. Struktura subkompleksu RecFR-DNA pokazuje ponadto sposób, w jaki jedna cząsteczka białka RecF oddziałuje z dwoma różnymi regionami pierścienia zbudowanego z czterech kopii białka RecR, co stabilizuje ten pierścień na cząsteczce DNA. Rekonstrukcje o niższej rozdzielczości subkompleksu RecR-RecO oraz całego układu RecFOR-DNA wyjaśniają, w jaki sposób białko RecO jest pozycjonowane, aby oddziaływać z ssDNA i białkiem SSB, co blokuje kompleks na styku ssDNA-dsDNA. Powyższe modele zostały zweryfikowane poprzez szereg eksperymentów biochemicznych i biofizycznych. Podsumowując, uzyskane wyniki integrują i wyjaśniają opublikowane dane biochemiczne dostępnych dla systemu RecFOR i budują podstawę do pełnego zrozumienia mechanizmu ścieżki rekombinacji homologicznej RecFOR.
Link do publikacji: https://www.nature.com/articles/s41594-023-00967-z
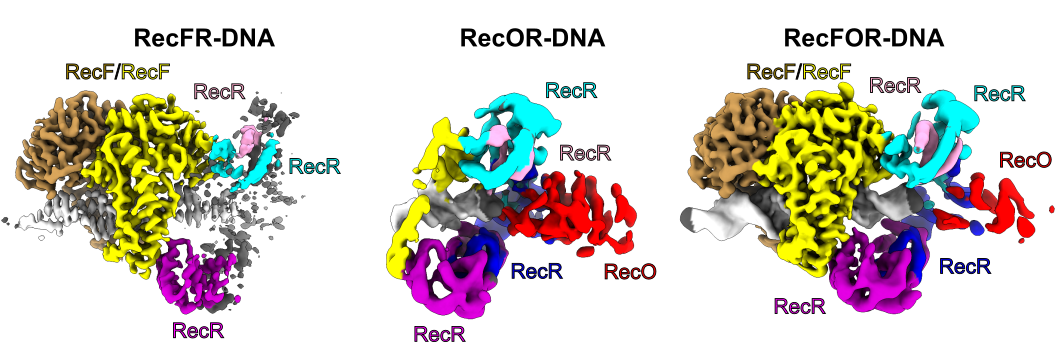
Fig: Rekonstrukcje kompleksu RecFOR-DNA oraz subkompleksów RecFR-DNA i RecOR-DNA wykonane przy użyciu kriomikroskopii elektronowej.
Dwumiesięczny staż w jednym z doskonale wyposażonych 14 laboratoriów MIBMiK, pod okiem wybitnych naukowców i ekspertów w swoich dziedzinach – taką ofertę programu stażowego uruchamia Międzynarodowy Instytut Biologii Molekularnej i Komórkowej w Warszawie.
Oferta jest skierowana do utalentowanych studentów nauk przyrodniczych, interesujących się obszarem biologii i biotechnologii. Podczas stażu wybrani studenci będą mieli możliwość uczestniczenia w projektach badawczych w laboratoriach MIBMiK pod kierownictwem znakomitych naukowców. Będzie to również doskonała okazja do pracy w międzynarodowych zespołach nad zagadnieniami związanymi z rozwojem współczesnej nauki, a także możliwość doskonalenia dotychczasowych umiejętności naukowych i analitycznych.
Dla najlepszych kandydatów Instytut oferuje miesięczny staż w pełnym wymiarze godzin z wynagrodzeniem w wysokości 2500 zł netto/mc. Jednostka zapewnia wstępne szkolenia naukowe i wsparcie ze strony doświadczonych naukowców i doktorantów oraz pełne wsparcie organizacyjne i administracyjne w przyjaznym i międzynarodowym środowisku pracy.
Studentów zainteresowanych ofertą zachęcamy do zapoznania się z tematyką badawczą poszczególnych laboratoriów i przesłania swojego zgłoszenia (CV i formularz rejestracyjny) na adres Ten adres pocztowy jest chroniony przed spamowaniem. Aby go zobaczyć, konieczne jest włączenie w przeglądarce obsługi JavaScript.
Zgłoszenia do programu stażowego przyjmowane są w trybie ciągłym. Do przesyłania aplikacji zapraszamy studentów studiów licencjackich lub magisterskich oraz osoby, które niedawno uzyskały tytuł zawodowy. Liczba miejsc jest ograniczona.
Z radością informujemy, że z początkiem kwietnia br. swoją działalność w Międzynarodowym Instytucie Biologii Molekularnej i Komórkowej w Warszawie rozpoczęły dwa nowe Laboratoria: Laboratorium Biofizyki Pojedynczych Cząsteczek, kierowane przez dr Ewelinę Małecką-Grajek oraz Laboratorium Genomiki Komórkowej, kierowane przez dr Aleksandrę Kołodziejczyk.
Badania realizowane w Laboratorium Biofizyki Pojedynczych Cząsteczek skupiają się na zrozumieniu najważniejszych procesów zachodzących w środowisku bakterii, w szczególności zależności i koordynacji w procesie celowania w RNA, degradacji i translacji u bakterii. Choć większość gatunków bakterii jest nieszkodliwa i często przynosi korzyści, w organizmach żywych występują również takie, które mogą wywoływać choroby zakaźne. W Laboratorium realizowane są również badania z wykorzystaniem metody mikroskopii TIRF z pojedynczą cząsteczką. Metoda ta pozwala na wizualizację setek biomolekuł unieruchomionych na szkiełku mikroskopowym, przez co każda cząsteczka może być analizowana oddzielnie.
Przedmiotem badań w Laboratorium Genomiki Komórkowej są mechanizmy molekularne leżące u podstaw przewlekłych procesów zapalnych towarzyszących włóknieniu narządów, zespołowi metabolicznemu i chorobom autoimmunologicznym. W Laboratorium badane są zależności między wątrobą a jelitem, ze szczególnym uwzględnieniem roli rezydującej tam mikrobioty. Identyfikowane są również zmiany w składzie bakterii i w fizjologii jelita wpływające na komórki wątroby. Prowadzone w Laboratorium badania mogą przybliżyć naukowców do uzyskania odpowiedzi na pytanie w jaki sposób zdrowie wątroby przyczynia się do homeostazy w jelicie.
Znamy już wyniki konkursu Best Papers Awards 2022! Nagrodzone zostały najlepsze prace naukowe afiliowane w MIBMiK, opublikowane w minionym roku. Do konkursu można było zgłosić każdą pracę eksperymentalną o charakterze naukowym, bez ograniczeń tematycznych. Najlepsze publikacje zostały wybrane przez Jury złożone z Liderów wszystkich Laboratoriów w MIBMiK. Głównym kryterium oceny były treść i znaczenie publikacji, a nie dane bibliometryczne. Liderzy nie mogli głosować na prace z własnego laboratorium. W 2022 roku nagrodzono następujące prace:
- 1 MIEJSCE
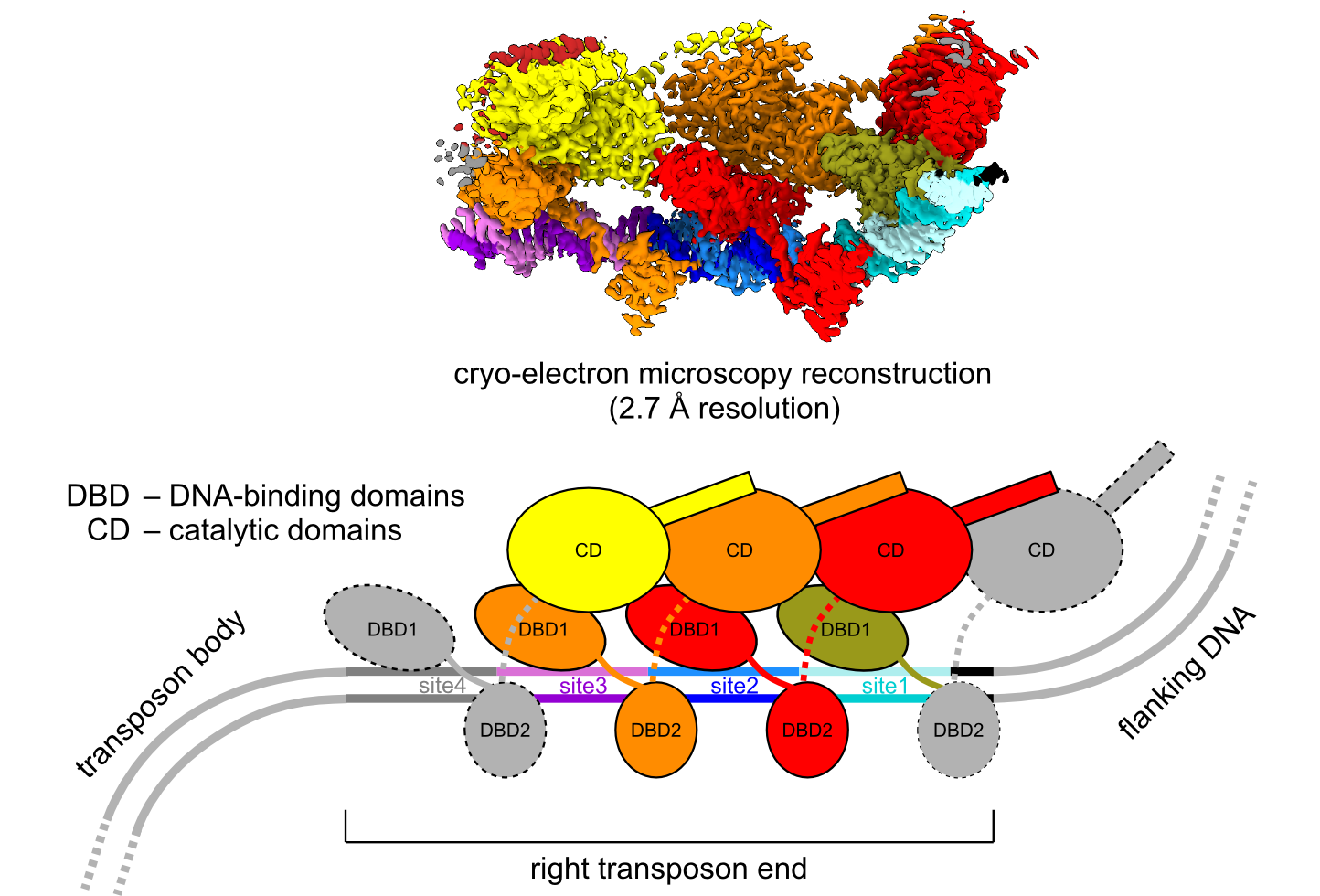
Kaczmarska Z*, Czarnocki-Cieciura M*, Górecka-Minakowska KM*, Wingo RJ, Jackiewicz J, Zajko W, Poznański JT, Rawski M, Grant T, Peters JE#, Nowotny M#. Structural basis of transposon end recognition explains central features of Tn7 transposition systems. Mol Cell, 2022; 82(14):2618-32.e7. doi: 10.1016/j.molcel.2022.05.005
*autorzy z równym wkładem; #autorzy korespondencyjni; pogrubioną czcionką zaznaczono nazwiska autorów z MIBMiK
Transpozony, zwane również „skaczącymi genami”, są fragmentami DNA, które mogą przemieszczać się w obrębie genomów lub pomiędzy nimi w procesie zwanym transpozycją. U bakterii transpozony uczestniczą w przekazywaniu między komórkami genów oporności na antybiotyki i genów wirulencji. Bakteryjny transpozon Tn7 należy do najlepiej zbadanych i najbardziej rozpowszechnionych transpozonów DNA. Przenoszenie Tn7 zachodzi przy udziale pięciu białek kodowanych przez ten element. Tn7 wykorzystuje mechanizm typu "wytnij-wklej", katalizowany przez heteromeryczną transpozazę TnsA-TnsB, która jest rekrutowana do docelowego DNA przez białko TnsC, będące ATPazą typu AAA+. TnsC oddziałuje z jednym z dwóch białek, które kierują transpozon do jego docelowej lokalizacji: TnsD lub TnsE. TnsD kieruje element do zachowanej w ewolucji sekwencji w chromosomie zwanej attTn7, natomiast TnsE umożliwia transpozycję do plazmidów koniugacyjnych. Traspozony związane z CRISPR (ang. CRISPR-associated transposon elements), które wykorzystują systemy CRISPR-Cas do transpozycji DNA kierowanej przez RNA, są spokrewnione z Tn7 i kodują transpozazy podobne do TnsB. Mogą one posłużyć do stworzenia nowych narzędzi do precyzyjnej edycji genów.
Naukowcy z Laboratorium Struktury Białka, kierowanego przez Marcina Nowotnego, we współpracy z grupą Joe Petersa z Cornell University, opisali strukturę i mechanizm działania prototypowej transpozazy TnsB z Tn7 E. coli. Badacze wykorzystali mikroskopię elektronową w reżimie kriogenicznym (cryo-EM) do określenia struktury białka TnsB w kompleksie z dwuniciowym DNA odpowiadającym prawemu końcowi transpozonu o rozdzielczości 2,7 Å. Struktura pokazuje, że kilka łańcuchów białka TnsB, które przyjmuje architekturę domen połączonych elastycznymi łącznikami, oddziałuje z powtarzającymi się miejscami wiązania TnsB w DNA. W wyniku tej interakcji domeny wiążące DNA i domeny katalityczne TnsB układają się naprzemiennie w spleciony sposób. TnsB tworzy niewiele sekwencyjnie specyficznych kontaktów z DNA, co prowadzi do preferencji sekwencyjnej, a nie do ścisłej specyficzności. Szereg cząsteczek TnsB, które wiążą się z wieloma słabo zachowanymi w ewolucji miejscami, które są położone w odpowiednich odstępach, zmienia tę preferencję w specyficzne rozpoznanie końca transpozonu. Naukowcy zaproponowali również model kompleksu białka TnsB z DNA po wycięciu transpozonu i wstawieniu go w nowe miejsce, co pozwala zrozumieć końcowe etapy reakcji prowadzonej przez Tn7 TnsB. Wyniki te pomagają wyjaśnić, w jaki sposób subtelne różnice w odstępach między miejscami wiązania są wykorzystywane do specyficznego rozpoznawania końca transpozonu. Opisane badania wyjaśniają najważniejsze cechy transpozonów Tn7.
- 2 MIEJSCE
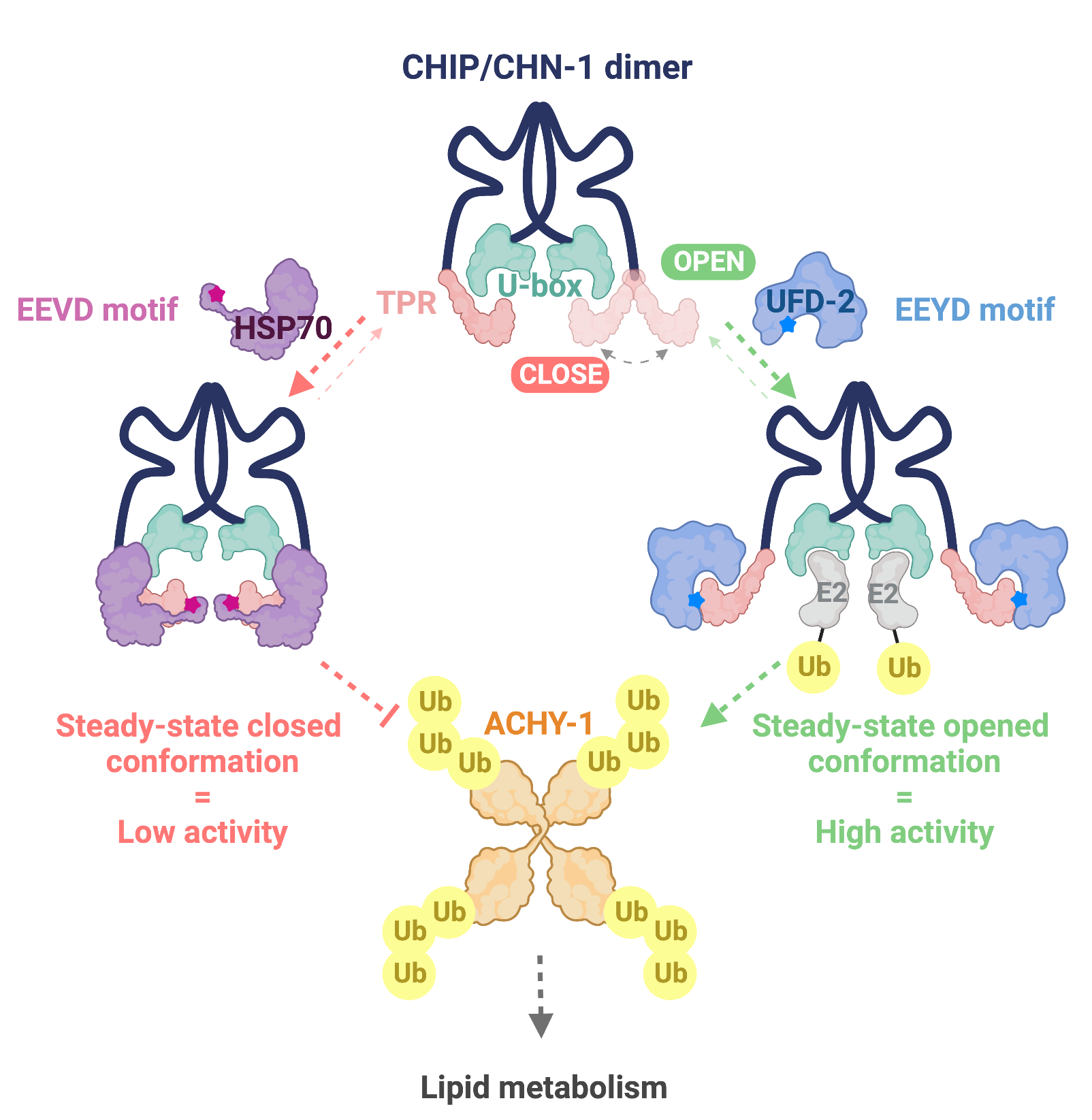
Das A, Thapa P, Santiago U, Shanmugam N, Banasiak K, Dąbrowska K, Nolte H, Szulc NA, Gathungu RM, Cysewski D, Krüger M, Dadlez M, Nowotny M, Camacho CJ, Hoppe T, Pokrzywa W#. A heterotypic assembly mechanism regulates CHIP E3 ligase activity. EMBO J, 2022; 41(15):e109566, doi: 10.15252/embj.2021109566
#autor korespondencyjny; pogrubioną czcionką zaznaczono nazwiska autorów z MIBMiK
Los białek eukariotycznych nadzorowany jest przez sieć białek opiekuńczych (chaperonów) i system ubikwityna-proteasom (UPS). Białko CHIP jest ważną dla kontroli jakości ligazą ubikwityny E3, która łączy system chaperonów z UPS w celu degradacji uszkodzonych białek. Pośredniczy ona również w ubikwitylacji niezależnej od chaperonów i może oddziaływać z innymi ligazami E3. Jednak regulacja działania CHIP i selektywności substratów w odpowiedzi na wiązanie do niego chaperonów i E3 pozostawała niejasna. Naukowcy z Laboratorium Metabolizmu Białek kierowanego przez Wojciecha Pokrzywę przeprowadzili strukturalno-funkcjonalną analizę kompleksu utworzonego przez CHIP i UFD-2, będącą również ligazą ubikwityny E3, kierując się ideą, że stworzą one wydajny system ubikwitylacji stanowiący alternatywę dla osi CHIP/chaperon. Uzyskane dane wykazały, że wiązanie UFD-2 promuje strukturalne wzmocnienie funkcji CHIP. Badacze wykazali również, że białko szoku cieplnego Hsp70 konkuruje z UFD-2 o wiązanie CHIP i negatywnie reguluje aktywność kompleksu poprzez stabilizację CHIP w nieaktywnym stanie. Wykorzystując nicienia Caenorhabditis elegans, naukowcy odkryli, że interakcja z UFD-2 umożliwia CHIP regulację S-adenozylohomocysteinazy, enzymu kluczowego dla metylacji w komórkach. Wyniki uzyskane przez grupę Wojciecha Pokrzywy otwierają nowe horyzonty w badaniach nad współpracą ligaz ubikwityny w uzyskiwaniu wysokiej aktywności i selektywności substratowej. Ponadto, ujawniony mechanizm przełączania procesywności CHIP ma potencjalne zastosowanie w technologii ukierunkowanej degradacji białek.
- 3 MIEJSCE
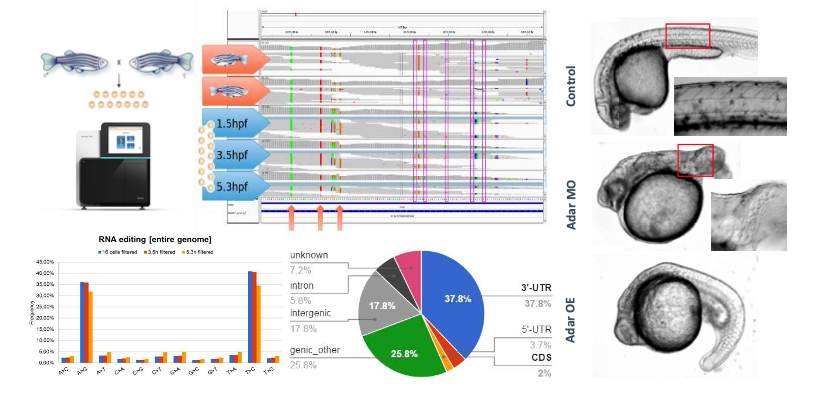
Niescierowicz K*, Pryszcz L*, Navarrete C*, Tralle E*, Sulej A*, Abu Nahia K, Kasprzyk ME, Misztal K, Pateria A, Pakuła A, Bochtler M#, Winata C#. Adar-mediated A-to-I editing is required for embryonic patterning and innate immune response regulation in zebrafish. Nat Commun, 2022; 13(1):5520, doi: 10.1038/s41467-022-33260-6
*autorzy z równym wkładem; #autorzy korespondencyjni; pogrubioną czcionką zaznaczono nazwiska autorów z MIBMiK
Konwersja adenozyny do inozyny (A-do-I) jest niezbędna do regulacji wrodzonego układu odpornościowego u ludzi i innych ssaków oraz jest związana z chorobami człowieka, w tym z chorobami autoimmunologicznymi. Enzym deaminaza adenozyny działająca na RNA (Adar) katalizuje edycję polegającą na deaminacji adenozyny (A) w pozycji C6 i przekształcenie jej w inozynę (I). Naukowcy z Laboratorium Genomiki Rozwoju Danio Pręgowanego i Laboratorium Biologii Strukturalnej, kierowani odpowiednio przez Cecilię Winatę i Matthiasa Bochtlera, badali rolę białka Adar u ryby danio, gdzie ulega ono wysokiej ekspresji w najwcześniejszych etapach embriogenezy. Śledzenie edycji RNA w całym genomie ryby poprzez połączone analizy genomu rodzicielskiego i transkryptomu embrionalnego ujawniła powszechną edycję A-do-I w matczynych i najwcześniejszych zygotycznych transkryptach, z których większość wystąpiła w rejonie 3' niepodlegającym translacji. Transkrypty, o których wiadomo, że odgrywają rolę w gastrulacji i kształtowaniu zarodka, zawierały wiele miejsc edycji, co sugeruje, że Adar może wywierać swoją funkcję za ich pośrednictwem. Eksperymentując z utratą i wzmocnieniem funkcji tego białka, badacze wykazali, że matczyny Adar z nienaruszoną domeną deaminazy jest konieczny do prawidłowego kształtowania się zarodka wzdłuż osi pionowej i strzałkowej. Analiza zygotycznych mutantów ujawniła odrębną funkcję białka Adar pochodzącego z zygoty w regulacji wrodzonej odpowiedzi immunologicznej, która to rola jest zachowana u ssaków. Podsumowując, badania wykazały udział edycji A-do-I przez Adar w kształtowaniu się zarodka i pokazały ewolucyjnie konserwowane znaczenie tego białka u ryb i ssaków.